
来源:特诺科研
前列腺癌多组学文章“没自测序数据、缺实验条件” ?怎么把“基因 - 疾病 - 药物” 的逻辑链讲得有说服力?福州医科大学及其附属医院的王玮团队(通讯作者 Wei Wang)最新发表在《Biological Procedures Online》的研究,直接给大家打了个 “低成本高产出” 的样 —— 靠 TCGA、GTEx、GWAS 等公共数据,加基于汇总数据的孟德尔随机化(SMR)、单细胞测序、分子对接等 “轻量级” 分析,就完整构建了 “易感基因→前列腺癌→候选药物” 的闭环,每一步都踩在医生发文的 “得分点” 上,今天咱们就扒透这篇文章的逻辑!
文章信息速览
原标题:Uncovering Novel Susceptible Genes and Therapeutic Targets of Prostate Cancer: a Multi-omics Study Integrating Summary-based Mendelian Randomization Analysis and Molecular Docking
期刊:Biological Procedures Online(IF=4.3)
关键词:前列腺癌、多组学分析、基于汇总数据的孟德尔随机化(SMR)、核心基因(ZNF217/BNIP2)、单细胞 RNA 测序(scRNA-seq)、分子对接、药物预测、表观遗传调控
这篇文章的核心逻辑是“公共数据打底→因果分析筛靶标→多维度验证→表观深挖→药物落地”,完全贴合医生 “缺时间、少实验资源” 的发文需求,具体可拆成 5 个关键步骤,每个步骤都标注了核心结果与对应图号,方便直接参考:
第一步:多源公共数据“搭框架”,奠定分析基础
这篇文章直接整合 3 类公共数据,从 “暴露(基因表达)” 到 “结局(前列腺癌风险)” 形成完整数据链:
暴露数据:用 eQTLGen 的血液 eQTL 数据(19250 个基因)和 GTEx 的前列腺组织特异性 eQTL 数据,锁定 “基因表达的遗传调控位点”;
结局数据:3 个独立前列腺癌 GWAS 数据集 ——PRACTICAL(发现队列)、UK Biobank(UKB)、FinnGen(验证队列),均为欧洲人群,样本量充足;
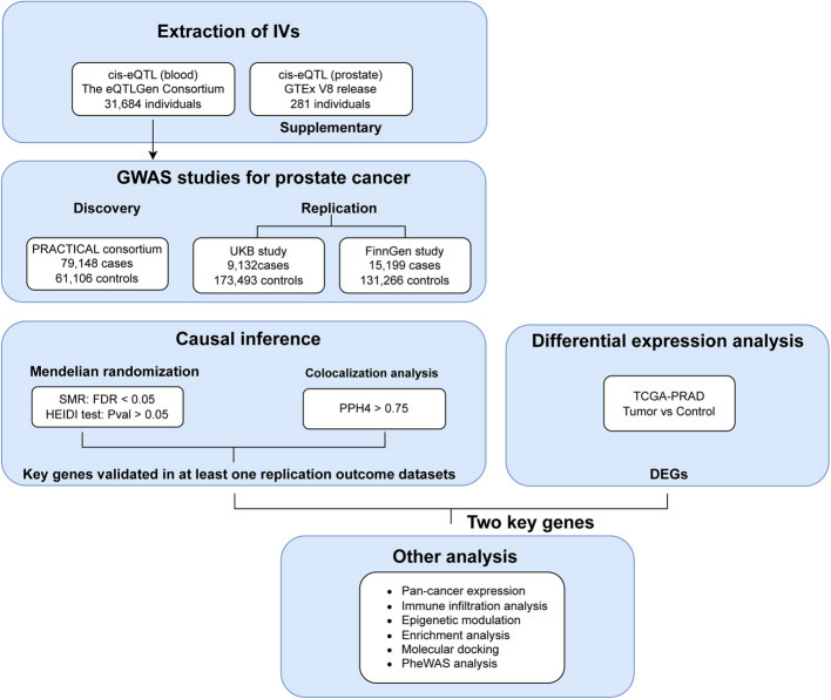
辅助数据:TCGA-PRAD 的 mRNA 测序数据、GTEx 的正常前列腺组织数据、GSE141445 的单细胞数据,用于后续验证。整个研究流程清晰(图 1),用公共数据覆盖 “遗传 - 转录 - 单细胞” 多个层面,既节省成本,又提升数据可信度。
第二步:SMR + 共定位 “去噪”,锁定 2 个核心因果基因
传统多组学容易陷在“基因 - 疾病关联” 的假阳性里,这篇文章用 “因果推断组合拳” 精准筛靶标,是医生发文的核心得分点:
SMR 分析:先通过 SMR+HEIDI 检验(排除连锁不平衡干扰),从血液 eQTL 中筛选出 15 个与前列腺癌有因果关联的基因(FDR<0.05,p_HEIDI>0.05),其中 SIK2 在 3 个 GWAS 中均验证(图 2a 森林图);
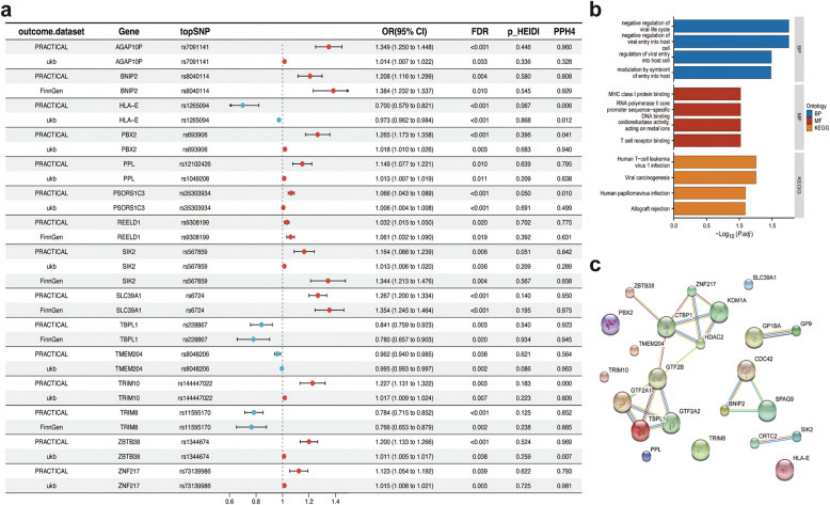
共定位验证:对 15 个基因做 Bayesian 共定位分析,挑出 PPH4≥0.75(共享同一因果变异)的 5 个基因(BNIP2、SLC39A1、ZNF217 等);
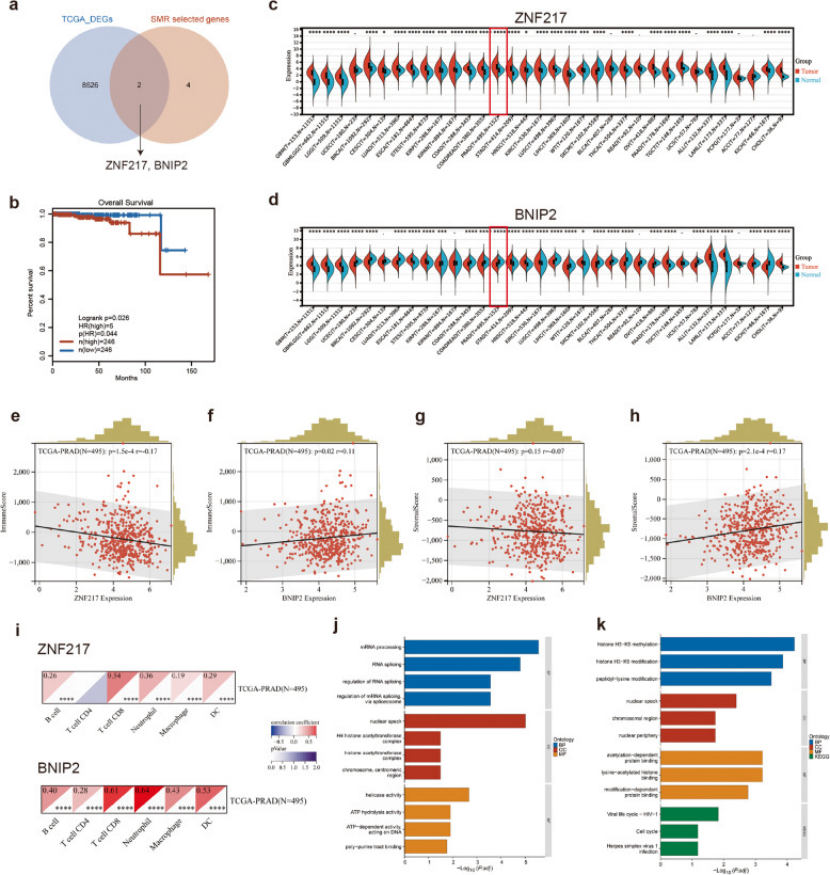
DEG 交集缩范围:把 15 个因果基因与 TCGA-PRAD 的差异表达基因(DEG)取交集,最终锁定ZNF217 和 BNIP2两个关键基因(图 3a Venn 图)—— 这一步既保证 “因果性”,又贴合 “疾病特异性表达”,让靶标更可靠。
第三步:从“生存预后” 到 “单细胞定位”,多维度验证核心基因
找到核心基因后,不能只停留在 mRNA 层面,这篇文章用 4 个维度做验证,让结果更扎实,医生发文可参考这种 “立体验证” 思路:
预后价值:两个基因的联合签名能预测前列腺癌患者生存期,高表达组预后更差(图3b Kaplan-Meier 曲线);
泛癌表达:ZNF217 在多数癌症(含前列腺癌)中高表达(p<0.0001),BNIP2 在前列腺癌中低表达(p<0.001),图 3c-d;
免疫浸润关联:ZNF217 高表达与免疫评分负相关,BNIP2 高表达与免疫 / 基质评分均正相关,且与 CD8+T 细胞、B 细胞等浸润正相关(图 3e-i);
单细胞定位:scRNA-seq 显示,BNIP2 主要在肿瘤微环境的内皮细胞(Endothelial_17 亚群)表达,ZNF217 主要在恶性上皮细胞表达(图 4f、h)—— 这一步用单细胞数据明确基因的 “细胞特异性”,让机制更具体。
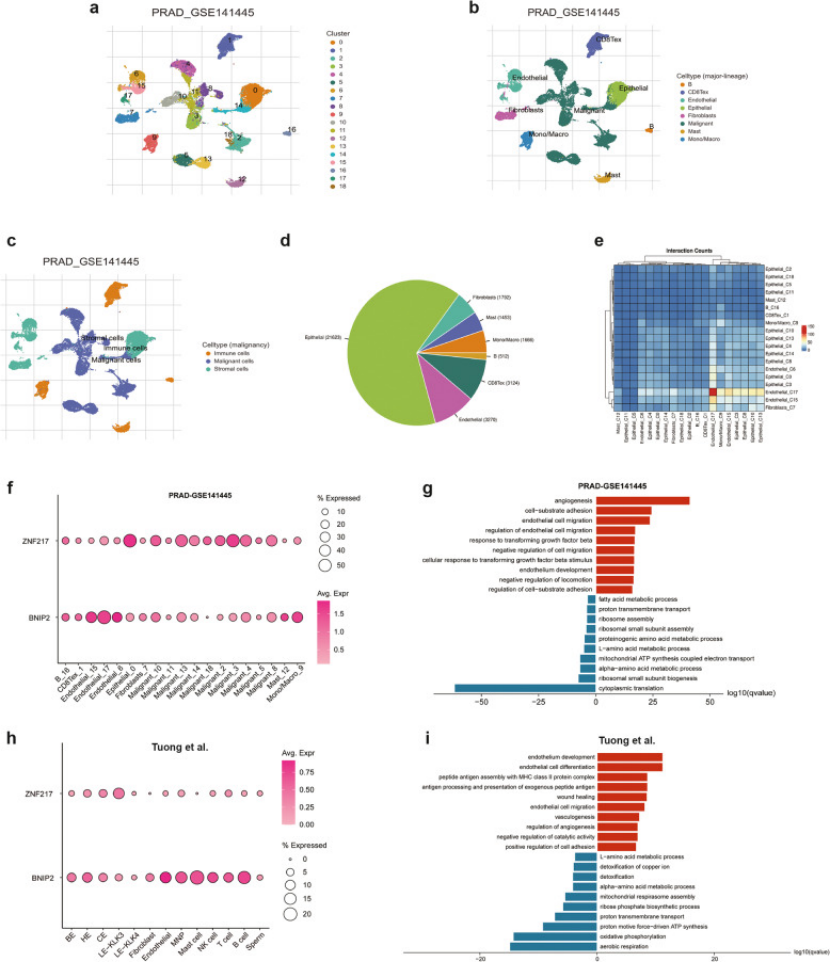
第四步:表观遗传深挖,补全“基因异常表达” 机制
医生发文要讲清“为什么基因会异常”,这篇文章通过表观分析补全逻辑链,关键结果带图号,说服力拉满:
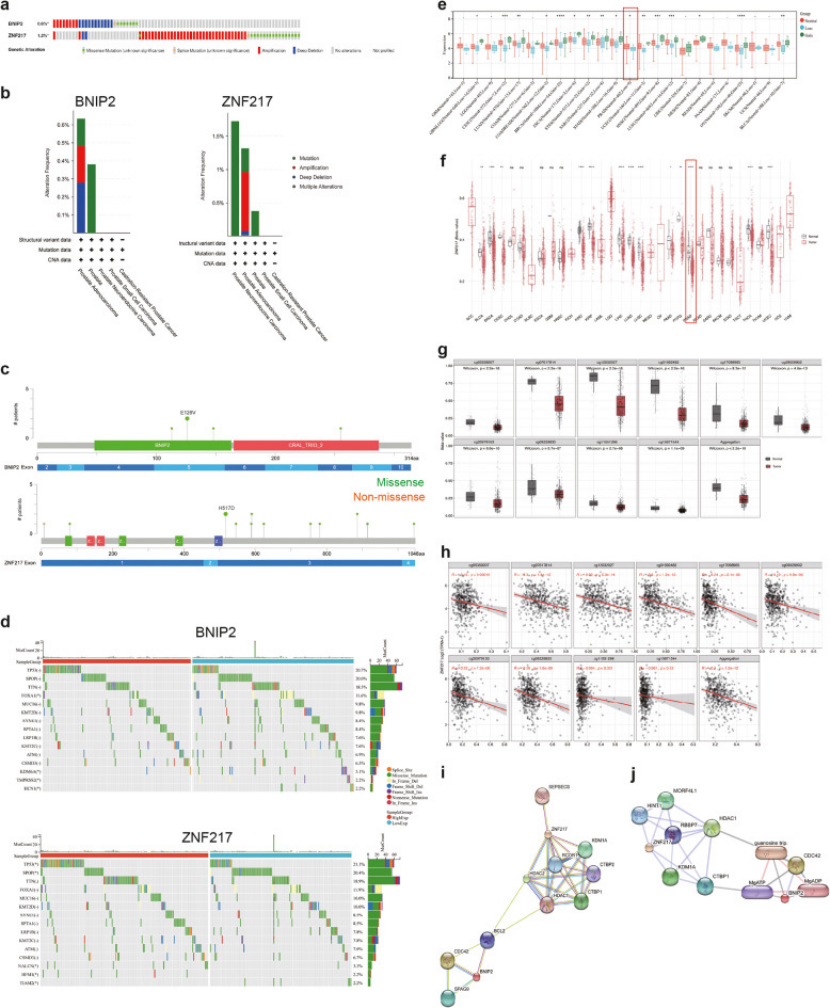
遗传变异:cBioportal 数据显示,ZNF217 在 1.2% 前列腺癌患者中存在扩增,BNIP2 在 0.6% 患者中存在深度缺失(图 5a-b);
拷贝数变异:BNIP2 低表达与拷贝数缺失相关,缺失组表达显著低于正常组(图 5e);
甲基化调控:ZNF217 在前列腺癌组织中呈低甲基化(p<0.0001),且 10 个 CpG 探针甲基化水平与表达负相关(图 5f-h)—— 明确 “ZNF217 高表达因低甲基化,BNIP2 低表达因拷贝数缺失”,机制更完整。
第五步:药物预测 + 分子对接,落地 “临床转化” 价值
多组学文章要贴近临床,这篇文章通过药物分析提升“转化意义”,是医生发文的加分项:
药物预测:用 Enrichr+DSigDB 筛选出 68 种潜在药物,优先选择 “未常规用于前列腺癌但已上市” 的 3 种 —— 多柔比星(DOX)、阿尔斯帕隆(alsterpaullone)、喜树碱(CPT);
分子对接验证:AutoDock Vina 对接显示,3 种药物与 ZNF217/BNIP2 均有强结合力(结合能 <-6.5 kcal/mol),比如多柔比星与 ZNF217 结合能 - 6.7 kcal/mol,与 BNIP2 结合能 - 7.3 kcal/mol(图 7a-f)—— 直接证明这 3 种药物可作为候选靶向药,让研究有临床应用潜力。
该文章的五个发文启示:
数据“贵精不贵多”:优先用多 cohort 公共数据(GWAS+TCGA + 单细胞),覆盖 “遗传 - 转录 - 细胞” 层面,比单数据集更易通过审稿;
因果分析“提档次”:加 SMR + 共定位,从 “关联分析” 升级为 “因果推断”,避免假阳性,审稿人更认可;
验证“多维度立体”:从预后→泛癌→免疫→单细胞,层层递进,每个验证都带数据和图号,结果更扎实;
机制“讲透异常原因”:补表观分析(甲基化、拷贝数),说清 “基因为什么异常”,逻辑链更完整;
落地“临床转化”:加药物预测 + 分子对接,让研究贴近治疗需求,提升文章价值。
看完这篇文章的拆解,是不是觉得前列腺癌多组学发文没那么难?不用烧钱做自测序,用好公共数据和“SMR + 验证 + 药物” 的逻辑链,就能把机制讲透!如果您在数据筛选、分析设计或文章撰写上遇到瓶颈,随时找我们 —— 从公共数据挖掘到因果分析,从图表绘制到文章润色,全流程帮您搞定多组学论文,让您高效发顶刊!
专注期刊投稿、发表十年,任何投稿、写作难题欢迎咨询!

PAPER INFORMATION

快速预审、投刊前指导、专业学术评审,对文章进行评价

校对编辑、深度润色,让稿 件符合学术规范,格式体例等标准
.png)
适用于语句和结构尚需完善和调整的中文文章,确保稿件达到要求
.png)
数据库包括: 期刊、文书籍、会议、预印章、书、百科全书和摘要等

让作者在期刊选择时避免走弯路,缩短稿件被接收的周期

根据目标期刊格式要求对作者文章进行全面的格式修改和调整
.png)
帮助作者将稿件提交至目标期刊投稿系统,降低退稿或拒稿率
.png)
按照您提供的稿件内容,指导完成投稿附信(cover letter)

北京总部:北京市海淀区碧桐园 3 号楼 2 层 211 广州办事处:广州市黄埔区科学城国际企业孵化器 E栋306 联系人:客服 / 18163670350
Copyright © 2022-2024 北京特诺科技有限公司 版权所有 备案/许可证编号为: 京 ICP 备 2023007944 号